
孙磊:博士,研究员,博士/硕士生导师,wnsr888威尼斯“齐鲁青年学者”
E-mail:sunlei0227@sdu.edu.cn
实验室主页:www.RNAlab.cn
教育背景:
2014.09 - 2019.10 清华大学 wnsr888威尼斯 生物信息学博士
2012.12 – 2013.06 乌普萨拉大学 生态中心 交换生
2010.09 - 2014.06 wnsr888威尼斯 泰山学堂 生物学学士
工作经历:
2022.12-至今 wnsr888威尼斯 wnsr888威尼斯 研究员
2019.11-2022.11 清华大学 wnsr888威尼斯 博士后
研究方向和成果:
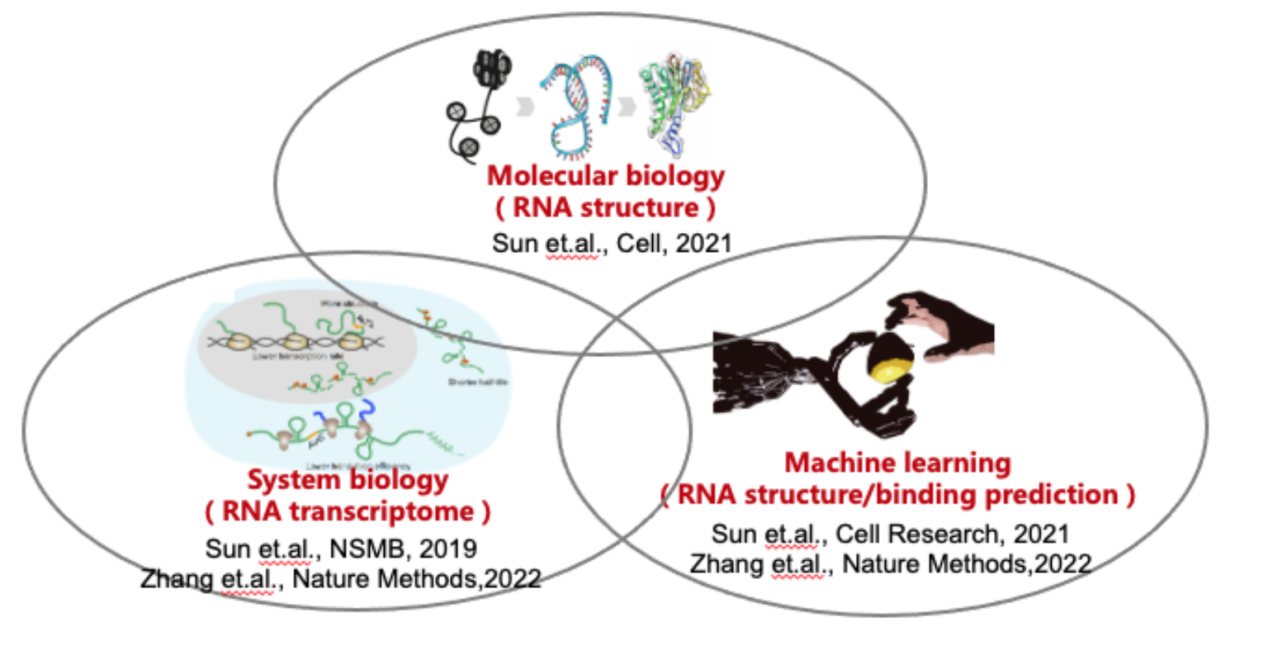
1. 技术开发方面
我们实验室对实验和计算方法的开发都很感兴趣。我们参与了许多技术的开发,包括亚细胞RNA结构测定(Sun et.al., NSMB, 2019 )和基于人工智能的RBP-RNA相互作用预测(Sun et.al., Cell Research, 2021)。我们还对单细胞RNA结构测定,共转录瞬时RNA结构测定等问题感兴趣。
2. 解决生物学问题方面
RNA结构和RBP-RNA相互作用在转录后调控中起着关键作用。我们可以通过icSHAPE、PrismNet、CLIP等技术从RNA结构和RNA-蛋白相互作用的角度研究病毒的调控网络,并通过靶向病毒的重要结构元件或相互作用的宿主蛋白来开发抗病毒药物(Sun et.al., Cell, 2021)。上述的研究思路,即整合高通量测序、深度学习和实验验证,为系统研究RNA结合蛋白的作用提供了一个可行且高效的思路。同样,我们也可以研究RBPs在斑马鱼发育过程中是如何调节基因表达的等其他科学问题。
3. 技术应用方面
基于之前的工作,我们已经确定了大量与RNA翻译、RNA降解等相关的保守RNA结构元件。这些RNA结构可以被利用来设计RNA分子,更好地帮助疫苗和RNA靶向药物的开发。
代表性论文(*为第一作者,#为通讯作者)
1.Xu, Y. *, J. Zhu*, W. Huang*, K. Xu, R. Yang, Q.C. Zhang#, and L. Sun#. PrismNet: predicting protein–RNA interaction using in vivo RNA structural information. Nucleic Acids Research, gkad353(2023)
2.Deng P.*, S.Q. Tan *, Q.Y. Yang, L. Fu, Y. Wu, H.Z. Zhu, L. Sun, …, Wang J.#, Liu J.#. tructural RNA components supervise the sequential DNA cleavage in R2 retrotransposon. Cell, 186(13):2865-2879.e20.(2023)
3.Zhang, J.*, Fei, Y.*, Sun, L.#, and Zhang, Q.C#. Advances and opportunities in RNA structure experimental determination and computational modeling. Nature Methods 19, 1193-1207.(2022)
4.Sun, L. *, Li, P. *, Ju, X. *, Rao, J. *, Huang, W. *, Zhang, S., Xiong, T., Xu, K., Zhou, X., Ren, L., et al. In vivo structural characterization of the whole SARS-CoV-2 RNA genome identifies host cell target proteins vulnerable to re-purposed drugs. Cell, 184, 1865-1883. (2021)
5.Sun, L. *, Xu, K. *, Huang, W. *, Yang, Y.T. *, Li, P., Tang, L., Xiong, T., and Zhang, Q.C. Predicting dynamic cellular protein-RNA interactions using deep learning and in vivo RNA structure. Cell Research, 31, 495-516. (2021)
6.Zhang, S., Huang, W., Ren, L., Ju, X., Gong, M., Rao, J., Sun, L., Li, P., Ding, Q., Wang, J., et al. Comparison of viral RNA-host protein interactomes across pathogenic RNA viruses informs rapid antiviral drug discovery for SARS-CoV-2. Cell Research. 4;1-15 (2021)
7.Sun, L.*, F. M. Fazal*, P. Li*, J. P. Broughton, B. Lee, L. Tang, W. Huang, E. T. Kool, H. Y. Chang# and Q. C. Zhang# "RNA structure maps across mammalian cellular compartments." Nature structural & molecular biology, 26 (4), 322-330 (2019).
8.Liu, Z. *, J. Wang*, H. Cheng*, X. Ke*, L. Sun, Q. C. Zhang and H. W. Wang "Cryo-EM Structure of Human Dicer and Its Complexes with a Pre-miRNA Substrate." Cell 173, 1191-1203 e1112, (2018).
9.Li, P. *, Y. Wei*, M. Mei*, L. Tang*, L. Sun, W. Huang, J. Zhou, C. Zou, S. Zhang, C. F. Qin, T. Jiang, J. Dai, X. Tan# and Q. C. Zhang# "Integrative Analysis of Zika Virus Genome RNA Structure Reveals Critical Determinants of Viral Infectivity." Cell Host Microbe 24, 875-886 e875, (2018).
10.Piao, M., L. Sun and Q. C. Zhang# "RNA Regulations and Functions Decoded by Transcriptome-wide RNA Structure Probing." Genomics Proteomics Bioinformatics 15, 267-278, (2017).
加入我们:
作为一个交叉实验室,我们既欢迎做湿实验的同学,也欢迎做分析的同学。同时,我们与“数学与交叉科学研究中心”联合招收具有数学、计算机研究背景的博士生。我们团队正在寻找对RNA结构、RNA调控、生物信息学分析和人工智能感兴趣的硕士、博士、博士后和研究助理。我们为我们的工作感到自豪,并努力做严谨的科学工作。
我们与清华大学张强锋实验室具有长期合作,你将有机会在清华大学进行联合培养。同时,你也将有机会与病毒学、胚胎发育、微生物和单细胞等方面的顶尖专家合作。
我的目标是为实验室学习的同学提供充分的指导、培训和支持,使你们能在学术界或工业界获得成功。我也希望实验室能够培养广泛的科学兴趣,因此更欢迎与wnsr888威尼斯的其他老师进行合作。
